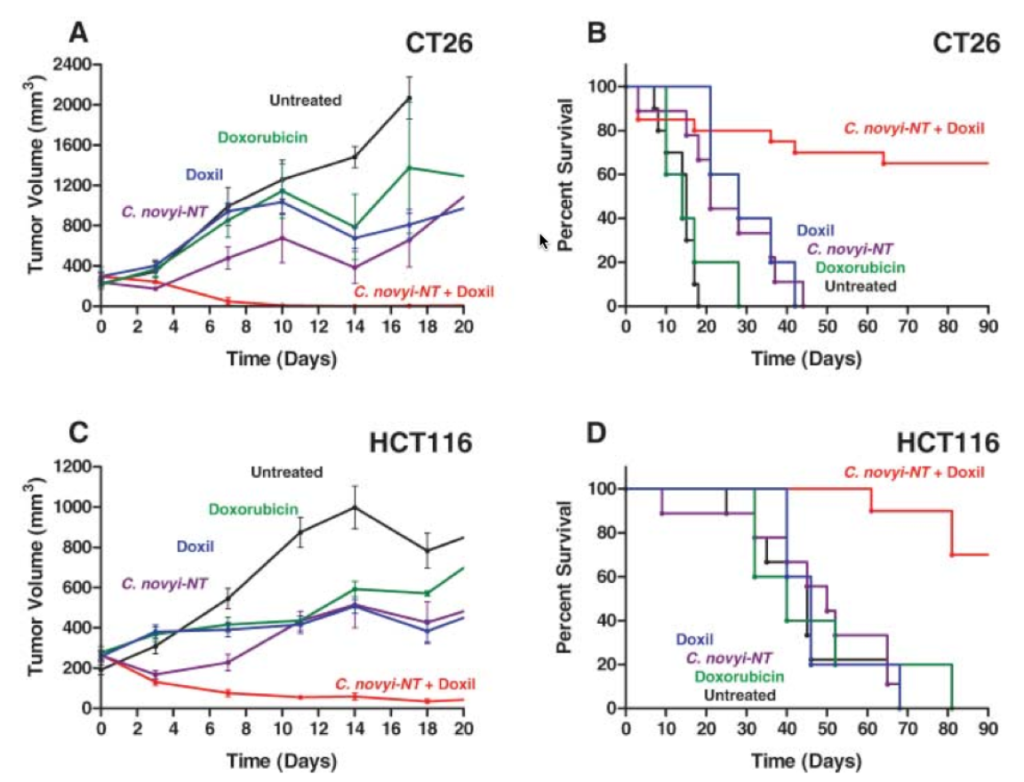
Esporas de un Bacillo indeterminado vistas a 100x Son las "bolitas" blancas, se ven así pues su pared no deja entrar a la tinción.
Bacillus subtilis es una bacteria común del suelo que forma una endospora cuando la comida escasea. No es patógena, aunque sus endosporas son muy resistentes sobreviviendo a muchos procesos de descontaminación. Pero lo que nos interesa es la formación de las endosporas.
En la naturaleza existen muchísimas formas de superar los periodos de escasez, la formación de endosporas es uno de ellos, está mediado por Spo0A un regulador maestro responsable de dar inicio a la proceso de formación de esporas. Hay muchísimos genes implicados en el proceso, un proceso largo y complejo, que implica duplicar el ADN, y producir un extra de estructuras celulares que con una pequeña cantidad de reservas de nutrientes sirvan para reactivarse cuando pase la mala época.

Podemos poner un ejemplo humano. En nuestro caso será el de una ciudad que suele vivir constantes ataques y catástrofes. Con el tiempo la gente ha aprendido, y todos poseen búnkers en el campo. Sin embargo no siempre pueden tenerlos habilitados. Preparar un búnker requiere varios días de trabajo, e irse a vivir al búnker sin que ocurra ninguna catástrofe implica que te despidan del trabajo (estás demasiado lejos para ir a la ciudad) y con ello morir de hambre.
El dilema no es ni mucho menos simple, la gente debe llevar maquinaria (proteinas), reparar las paredes y estructuras del búnker (septo separador), llevar una copia de sus datos personales (copia del genoma) y por supuesto algo de comida. La cosa se complica más aún si os digo que nunca se sabe a ciencia cierta cuando va a ocurrir la catástrofe. Todo son comentarios y habladurías por el barrio.
"-Ayer vi un avión...eso es que se avecina bombardeo"
"-El otro día el viento parecía muy calido, mi abuelo siempre decía que 'viento caliente tifón en la frente"
"-He ido al super, y el dueño estaba llevándose todas las patatas fritas, ¡ es señal de escasez !"
Ante tantos rumores nuestros ciudadanos nunca saben qué hacer, algunos no se creerán nada, y cuando llegue la catástrofe les pillará por sorpresa, otros muy asustadizos se irán al búnker a la primera de cambio, y morirán allí de hambre, o saldrán cuando esté la cosa ardiente.
¿Cómo logra solucionar este problema nuestra bacteria? Bacillus subtilis, hace algo inteligente y que a nuestros ciudadanos de la metáfora anterior nunca se les habría ocurrido. ¡ Encuestas !
Mediante procesos, seguramente de
QS, las bacterias consultan continuamente las señales de toda la comunidad, pero no es tan fácil...las comunidades de
B. subtilis son de miles de millones, el ruido es espantoso...
Existe un gran problema de muestreo estadístico. Si la bacteria espera una señal clara, precisa y segura de que el alimento se ha terminado y debe formar la endospora,
ya no tendrá tiempo de hacerlo. La decisión de formarla debe ser tomada antes de estar segura al 100%.
Lo que parece que sucede es que
B. subtilis comienza la preparación de la endospora incluso mientras no está decidida a formarla y mediante un sistema de "
feed forward", ¡
realiza promedios de las señales que le llegan durante un largo periodo de tiempo ! Pudiendo en cualquier momento si retrasar, parar o acelerar la formación de la endoespora. Esto le permite tomar decisiones precisas en base una media de las señales que le llegan, sin retrasarse ni dejarse influir por el ruido general de la colonia.
A mi personalmente me parece asombroso....
 |
| Colonia de B. subtilis foto por M. Fujita/UH PNAS |
En la foto podemos ver una colonia de Bacillus subtilis, se usaron marcadores fluorescentes para mostrar la expresión de genes que forman esporas. Podemos ver bacterias en todos los procesos de la esporulación, y sin embargo el medio es el mismo para todas y los nutrientes también. Podemos ver no esporulantes (azul oscuro casi verde) y las que están empezando a formar esporas (amarillas y la zona de la endospora en rosa)
 Narula, J., Devi, S., Fujita, M., & Igoshin, O. (2012). PNAS Plus: Ultrasensitivity of the Bacillus subtilis sporulation decision Proceedings of the National Academy of Sciences DOI: 10.1073/pnas.1213974109
Narula, J., Devi, S., Fujita, M., & Igoshin, O. (2012). PNAS Plus: Ultrasensitivity of the Bacillus subtilis sporulation decision Proceedings of the National Academy of Sciences DOI: 10.1073/pnas.1213974109
1 Persona especializada en asuntos concernientes a la dirección de los Estados o instruida en materias de política
2 Jefe de un Estado.
3 Persona que se dedica a la estadística. estadístico.
Diccionario Manual de la Lengua Española Vox. © 2007 Larousse Editorial, S.L.
AMPLIACIÓN.
Ampliamos la entrada con la foto con total detalle de una
espora de
B. subtilis.
 |
| Créditos de fotografía: Patrick Eichenberger, New York University (NIH/NIGMS Galería de imágenes) |
Podemos ver las cuatro capas que componen la protección de la espora, y que permiten sobrevivir situaciones ambientales que matarían normalmente a las bacterias. Estas situaciones incluyen temperatura alta, irradiación UV intensa, desecación, daño químico y la acción enzimática.
Pero no sólo os traigo esta foto cortesía de la ASM, además os traigo un vídeo en el que se muestra en directo la acción de los genes que responden al estrés. En él podemos ver como estos genes están siempre activos, pero varían su intensidad si la situación empeora dándole a la bacteria la capacidad de estar siempre alerta pero dejándole hacer mientras tanto vida normal.
Michael Elowitz, Caltech University Pulsating response to stress in bacteria
Al unir proteínas fluorescentes al circuito genético responsable de la respuesta a estrés, los investigadores pueden observar el uso de estos genes como destellos verdes. Este video se muestran células multiplicándose a lo largo de más de 12 horas.
En respuesta a un ambiente de estrés por falta alimento carente, B. subtilis activa un conjunto de genes que le ayudan a responder a las dificultades. En lugar de activar esos genes sólo cuando la comida falta como se pensaba anteriormente, los investigadores descubrieron que las bacterias activan y desactivan continuamente estos genes, aumentando la frecuencia de estos pulsos cuando la cosa empeora.